728x90
반응형
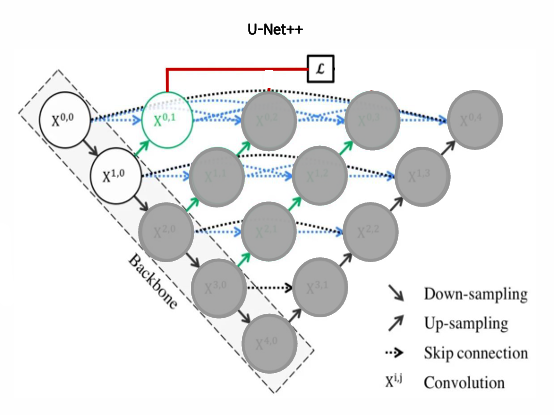
UNet3+는 전통적인 UNet 모델의 한계를 극복하고자 제안된 세분화 모델이다. 특히 의료 이미지와 같은 고해상도 데이터에서 더욱 정교한 경계와 세부 정보를 보존하기 위해 설계되었다. 이 모델은 다중 스킵 연결(Multi-scale Skip Connection)과 복합 기능 집계(Deep Supervision)를 활용해 기존 UNet보다 뛰어난 성능을 보인다.

UNet3+의 구조적 특징
- 다중 스킵 연결 (Full-scale Skip Connection)
UNet3+는 다양한 스케일의 정보를 병합하는 독창적인 스킵 연결을 도입했다. 기존 UNet은 단순히 인코더와 디코더 간의 동일한 레벨에서만 스킵 연결을 사용했지만, UNet3+는 더 많은 스케일의 피처 맵을 동시에 활용한다.
이를 통해 지역적 세부 정보와 전역적 문맥 정보를 효과적으로 통합할 수 있다. - 디코더 구조
각 디코더 레벨에서 인코더의 모든 스케일과 연결되어 있다. 이러한 구조는 인코더의 다양한 해상도 정보를 최대한 활용하며, 다중 스케일의 피처를 통합하여 디코더에서 학습한다. 이러한 통합은 모델이 정교한 경계를 학습하도록 돕는다. - Deep Supervision
Deep Supervision은 각 디코더 레벨에서 중간 결과를 생성하고 이를 학습에 활용하는 방법이다. 이를 통해 모델의 학습 안정성과 수렴 속도를 높이고, 다양한 레벨의 예측 결과를 결합하여 최종 출력을 생성한다. - 파라미터 효율성
UNet3+는 기존 UNet 대비 구조가 복잡해졌음에도 불구하고, 파라미터 수가 크게 증가하지 않아 경량화된 환경에서도 사용 가능하다. 이는 실제 의료 이미지 분석과 같은 고효율이 요구되는 작업에 특히 유리한 특징이다.
UNet3+의 장점
- 경계 학습 강화
다양한 스케일의 정보를 통합하여 더욱 선명하고 정확한 경계를 학습할 수 있다. - 효율적인 학습
Deep Supervision 기법을 통해 학습을 빠르게 진행하며, 각 단계의 중간 결과를 활용해 모델의 안정성을 높인다. - 일반화 성능 향상
여러 스케일의 피처 맵을 통합하여, 다양한 데이터셋에서도 우수한 성능을 보인다.
UNet3+의 활용 사례
- 의료 영상 분석
의료 영상 데이터는 고해상도의 정교한 세분화를 요구한다. UNet3+는 특히 X-ray, CT, MRI 등에서 장기나 병변의 정밀한 경계를 학습하는 데 적합하다. - 위성 이미지 분석
UNet3+는 위성 이미지에서 지형이나 건물 경계를 정확히 분할하는 데 활용된다. - 생물학적 이미지 처리
현미경 데이터를 분석할 때, 세포나 조직 경계의 세분화에도 UNet3+가 효과적이다.
UNet3++을 PyTorch로 구현한 코드
import torch
import torch.nn as nn
import torch.nn.functional as F
class ConvBlock(nn.Module):
"""
기본 Convolution Block: 2개의 Conv2D + BatchNorm + ReLU 연산을 수행.
"""
def __init__(self, in_channels, out_channels):
super(ConvBlock, self).__init__()
self.conv1 = nn.Conv2d(in_channels, out_channels, kernel_size=3, padding=1)
self.bn1 = nn.BatchNorm2d(out_channels)
self.conv2 = nn.Conv2d(out_channels, out_channels, kernel_size=3, padding=1)
self.bn2 = nn.BatchNorm2d(out_channels)
self.relu = nn.ReLU(inplace=True)
def forward(self, x):
x = self.relu(self.bn1(self.conv1(x)))
x = self.relu(self.bn2(self.conv2(x)))
return x
class UNet3PlusPlus(nn.Module):
"""
UNet3++ 모델 구현.
- 인코더-디코더 구조와 다중 스킵 연결을 통해 다양한 스케일 정보를 통합.
"""
def __init__(self, in_channels=3, out_channels=1, base_channels=64):
super(UNet3PlusPlus, self).__init__()
# 인코더 정의
self.enc1 = ConvBlock(in_channels, base_channels)
self.enc2 = ConvBlock(base_channels, base_channels * 2)
self.enc3 = ConvBlock(base_channels * 2, base_channels * 4)
self.enc4 = ConvBlock(base_channels * 4, base_channels * 8)
self.enc5 = ConvBlock(base_channels * 8, base_channels * 16)
# 디코더 정의
self.dec4 = ConvBlock(base_channels * (16 + 8), base_channels * 8)
self.dec3 = ConvBlock(base_channels * (8 + 4), base_channels * 4)
self.dec2 = ConvBlock(base_channels * (4 + 2), base_channels * 2)
self.dec1 = ConvBlock(base_channels * (2 + 1), base_channels)
# 마지막 출력
self.final_conv = nn.Conv2d(base_channels, out_channels, kernel_size=1)
# 최대 풀링
self.pool = nn.MaxPool2d(kernel_size=2, stride=2)
def forward(self, x):
# 인코더 단계
e1 = self.enc1(x)
e2 = self.enc2(self.pool(e1))
e3 = self.enc3(self.pool(e2))
e4 = self.enc4(self.pool(e3))
e5 = self.enc5(self.pool(e4))
# 디코더 단계
d4 = self.dec4(torch.cat([F.interpolate(e5, scale_factor=2, mode='bilinear', align_corners=True), e4], dim=1))
d3 = self.dec3(torch.cat([F.interpolate(d4, scale_factor=2, mode='bilinear', align_corners=True), e3], dim=1))
d2 = self.dec2(torch.cat([F.interpolate(d3, scale_factor=2, mode='bilinear', align_corners=True), e2], dim=1))
d1 = self.dec1(torch.cat([F.interpolate(d2, scale_factor=2, mode='bilinear', align_corners=True), e1], dim=1))
# 최종 출력
out = self.final_conv(d1)
return out
# 모델 테스트
if __name__ == "__main__":
# 입력 텐서 크기 (배치 크기, 채널 수, 이미지 크기, 이미지 크기)
x = torch.randn((1, 3, 256, 256))
# 모델 정의
model = UNet3PlusPlus(in_channels=3, out_channels=1)
print(model)
# 출력 확인
output = model(x)
print("Output shape:", output.shape)
코드 설명
- ConvBlock:
- 기본적으로 2개의 Conv2D와 BatchNorm, ReLU를 포함.
- UNet3++의 인코더와 디코더에서 사용.
- 인코더:
- 입력 이미지를 단계적으로 다운샘플링하며 다양한 스케일의 피처를 추출.
- 디코더:
- 다중 스케일의 정보를 병합하기 위해 torch.cat과 F.interpolate을 사용하여 업샘플링 및 피처 결합.
- 최종 출력:
- 마지막에 1x1 Convolution을 사용하여 원하는 클래스 수로 출력.
- 테스트:
- 모델을 정의하고, 임의 입력 데이터를 통해 출력 크기를 확인.
이상입니다. 끝!
감사합니다.
728x90
반응형
'딥러닝 (Deep Learning) > [03] - 모델' 카테고리의 다른 글
| Scikit-Learn 모델 분류 및 개념 정리 (0) | 2025.04.03 |
|---|---|
| Maskformer (0) | 2024.11.27 |
| DeepLab v1 아키텍쳐 분석 (1) | 2024.11.20 |
| SegNet의 아키텍처 (3) | 2024.11.18 |
| SVM(Support Vector Machine) 이란? [R-CNN] (0) | 2024.09.03 |